Herzlich willkommen am Institut für angewandte Medizininformatik
Das Institut für angewandte Medizininformatik (IAM) gehört zum Zentrum für Experimentelle Medizin des Universitätsklinikums Hamburg-Eppendorf.
Das Beginn 2021 neu geschaffene Institut führt komplexe, medizinische Daten zusammen und erzeugt in Partnerschaft mit Expert:innen effektiv gegen Krankheiten nutzbares Wissen, welches zu einem tieferen Krankheitsverständnis und zu einer erfolgreicheren Behandlung von Patienten führt. Ein agil arbeitendes Team erschafft und publiziert innovative Prozesse sowie maßgeschneiderte Methoden und Werkzeuge für Ärzt:innen und Forscher:innen. Unser Ziel ist es, der medizinischen Forschung heterogene Datenbestände zu erschließen, interdisziplinäre und internationale Kooperationen zu ermöglichen und komplexe Analysen zu vereinfachen.
Wir bieten Studierenden verschiedener (MINT) Studiengänge an Abschlussarbeiten bei uns am Institut zu schreiben. Mehr Infos zu den Abschlussarbeiten finden Sie hier.
Institutsdirektor
Anfangs lehrte Professor Ückert in Münster als Juniorprofessor und dann in Mainz als Universitätsprofessor und Leiter der Medizininformatik. Dabei wurde die Unterstützung und oft auch erst Initialisierung der medizinischen Verbundforschung für ihn zu einem Schwerpunkt. Ein Beispiel für einen komplexen Verbund ist das Deutsche Konsortium für translationale Krebsforschung (DKTK), eines der vom BMBF geförderten Deutschen Zentren der Gesundheitsforschung. Für die Entwicklung von dessen IT-Plattform, die alle elf Standorte des DKTK verbindet und gemeinsame Forschung mit den Daten und Biomaterialien ermöglicht, war er verantwortlich. Als ein Bestandteil eines „Spitzenclusters“ (Hightech-Strategie des BMBF) entwickelte Professor Ückert zudem ein internetbasiertes Portal, um verschiedene Omics-Daten kombiniert mit klinischen Daten teilautomatisiert biometrisch auswerten zu können.
Ab Januar 2016 leitete er als Universitätsprofessor der Universität Heidelberg die Medizinische Informatik in der Translationalen Onkologie am Deutschen Krebsforschungszentrum. Hier wurden hochkomplexe Daten in Partnerschaft mit Experten aus verschiedenen Disziplinen in Wissen umgewandelt, das zu Diagnose- und Therapiezwecken eingesetzt werden kann. Die in dem zusammen mit SAP entwickelten Data-Warehouse gesammelten Daten werden automatisch mit Informationen aus Publikationen und aus verschiedenen Datenbanken kombiniert. Über die Kenntnis von Modellen, aber auch mittels Algorithmen des maschinellen Lernens, wurden klinische Entscheidungshilfen gegeben und Forschungshypothesen generiert, um zu einem tieferen Verständnis der Erkrankung beizutragen.
Die Forschungsgruppen
Abschlussarbeiten
Am IAM bieten wir Studierenden diverser (MINT) Studiengänge die Möglichkeit, Projekt-, Bachelor-, Masterarbeiten sowie medizinische Promotionen bei uns zu verfassen. Die verfügbaren Themen finden Sie untenstehend. Bei Interesse senden Sie bitte eine E-Mail an die jeweilige Ansprechperson und geben das für Sie interessante Thema im Betreff an. Alle aufgeführten Themen sind derzeit noch verfügbar.
-
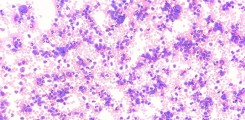
Zellklassifikation
Studienarbeit: Zellklassifikation (Bachelor/Projekt/Master)
Kollaboration von KI und Expert:innen:
Human-in-the-loop-Systeme zur effizienten Segmentierung und Klassifikation von Blut- und Knochenmarkszellen.Blut- und Knochenmarksausstriche spielen in der Diagnostik von Leukämie eine entscheidende Rolle. Aus der Verteilung der Zellbestandteile lassen sich wichtige diagnostische Rückschlüsse auf das Vorhandensein und das Fortschreiten der Erkrankung ziehen. Bis dato erfolgt diese Evaluation manuell durch klinische Expert*innen. Ziel der Arbeit ist das Entwickeln eines prototypischen Tools zur semiautomatischen Analyse von Blut- und Knochenmarksausstrichen und ein automatisches Update der zugrunde liegenden KI-Modelle durch intelligente Integration neuer Datenpunkte.
Aufgaben:
- Segmentierung von Einzelzellbildern aus Knochenmark- und Blutausstrichen mittels
neuronaler Netze - Annotation der Daten durch CVAT
- Online-Finetuning von „lightweight“ KI-Klassifikatoren auf Basis erlernter Zellrepräsentationen
Voraussetzungen:
- Technischer Studiengang wie Informatik/ Physik/ (Medizin-) Ingenieurwesen o.ä.
- Grundkenntnisse im (objekt-orientierten) Programmieren mit Python
- Gerne erste Erfahrungen mit Deep (pytorch), Machine Learning (scikit-learn) & Softwareentwicklung
Ansprechpartner:
Prof. Dr. René Werner,
gerne formlos an r.werner@uke.de
Image Processing and Medical Imaging Group - Segmentierung von Einzelzellbildern aus Knochenmark- und Blutausstrichen mittels
-
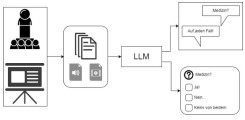
LLM für medizinische Sekundärdaten
Studienarbeit: LLM für medizinische Sekundärdaten (Bachelor/Projekt/Master)
LLM-Einsatz zur Generierung von Queries für die Kohortenselektion von medizinischen Sekundärdaten
Seit Oktober 2022 ist es am UKE möglich, mittels des Datenhotels medizinische Sekundärdaten aus verschiedensten Datenquellen zu verbinden und ausgeben zu lassen, um diese für die Auswertung und Hypothesentestung nutzen zu können. Die initiale Kohortenselektion ist dabei ein aufwendiger, aber kritischer Schritt, um die spätere Anfragen- und Datenqualität zu gewährleisten.
In diesem Projekt sollen mittels LLMs oder anderen Architekturen aus einer natürlichsprachlichen Beschreibung der gewünschten Kohorten mit interaktivem Feedback des Nutzers die technische und menschlich lesbare Anfrage generiert werden, die dann weitergehend für die Verarbeitung im Datenhotel genutzt werden kann.Aufgaben:
- Einsatz von LLMs zur Umwandlung von natürlichsprachlicher Eingabe in strukturierte Queries, die es ermöglichen, Daten aus dem Datenhotel zu extrahieren
- Einbindung von RAGs (Retrieval Augmented Generation) in das Modell, um Korrektheit der Anfrage zu erhöhen
- Entwicklung einer Pipeline mit menschlichem Feedback zur Verbesserung der Anfragenparameter
- Validierung der Ergebnisse anhand statistischer und „Utility“ Metriken (Fragestellungen auf den Datensätzen)
Voraussetzungen:
- Technischer Studiengang wie (Medizin-) Ingenieurwesen / Physik / Bioinformatik / Informatik oder ähnliches
- Grundkenntnisse Jupyterlab und Python
- Wünschenswert: Erste Erfahrungen mit dem Einsatz von LLMs
Ansprechpartnerin:
Dr. Layla Tabea Riemann
gerne formlos per E-Mail an: l.riemann@uke.de
Knowledge Integration in Precision Medicine -
Transformer
Studienarbeit: Transformer (Bachlor/Projekt/Master)
Effiziente Videoanalyse in der medizinischen Bildgebung mittels Transformer-Architekturen und Self-supervised Learning
Videos aus der medizinischen Bildgebung, beispielsweise wie Laryngoskopien und angiografische Aufnahmen, stellen eine große Herausforderung für die automatische Bildverarbeitung dar. Annotationen, zum Beispiel für die Beurteilung der Intubierbarkeit eines Patienten oder des Schweregrads eines Gefäßverschlusses, nur für bestimmte Abschnitte des Videos oder als globales Label für das gesamte Video verfügbar. Darüber hinaus führen Hardwarebeschränkungen zu langsamen und aufwändigen Trainingsprozessen.
Das Ziel dieses Projekts ist es, durch geschicktes Vortrainieren und den Einsatz von Transformer-Architekturen verschiedene Klassifikationsszenarien zu untersuchen. Dazu soll eine Trainingspipeline entwickelt werden, die es erlaubt Modelle Annotations und Ressourcen effizient, auf neue Fragestellungen zu adaptieren.Aufgaben:
- Einsatz und Anpassung von Transformer-Architekturen zur Kombination der einzelnen Videoframes (bzw. deren erlernte Repräsentationen) für verschiedene Klassifikationsszenarien
- Weiterentwicklung der Trainingspipeline für neue Fragestellungen
- Validierung der Ergebnisse anhand von zwei klinischen Datensätzen: Angiographien & Laryngoskopien
Voraussetzungen:
- Technischer Studiengang wie Informatik/ Physik/ Bioinformatik/ (Medizin-) Ingenieurwesen o.ä.
- Grundkenntnisse im (Objekt-orientierten) Programmieren mit Python
- Gerne erste Erfahrungen mit Deep (pytorch) & Machine Learning (scikit-learn)
Ansprechpartner:
Prof. Dr. René Werner,
gerne formlos an r.werner@uke.de
Image Processing and Medical Imaging Group -
LLM in der medizinischen Lehre
Studienarbeit: LLM in der medizinischen Lehre (Bachelor/Projekt/Master)
LLM-Einsatz zur Generierung von Studienmaterialien in der Lehre in der Medizin
Lehre stellt einen der Grundpfeiler des Universitätsklinikums Hamburg-Eppendorf dar. In einem sich stets und rapide weiterentwickelndem Feld wie der Medizin stehen die Vorlesungen selbst vor der Herausforderung fortlaufend aktuell und zugänglich zu bleiben, was sowohl Lehr- als auch Studienmaterial angeht. Dies stellt einen enormen Aufwand für die Lehre dar.
In diesem Projekt sollen LLMs zur Generierung von Studienmaterialien, vor allem zum Erstellen von Multiple-Choice-Aufgaben, aus den vorhandenen Lehrmaterialien verwendet, weiterentwickelt und evaluiert werden. Die Materialien sind nicht beschränkt auf Folien und Textdokumenten, sondern können auch Audioaufnahmen umfassen. Ziel ist es, den Studierenden ein kontinuierliches Lernen anhand des Curriculums zu ermöglichen.Aufgaben:
- Aufsetzen eines Systems zur automatischen Einbindung von Lernmaterialien in vorhandene LLMs
- Möglich: Einsatz von LLMs zur interaktiven Nutzung und Recherche in vorhandenen Lehrmaterialien (Chatbot)
- Möglich: Generierung von Lernkarten / Multiple Choice Fragebögen
Voraussetzungen:
- Technischer Studiengang wie (Medizin-) Ingenieurwesen / Physik / Bioinformatik / Informatik oder ähnliches
- Grundkenntnisse Jupyterlab und Python
- Wünschenswert: Erste Erfahrungen mit dem Einsatz von LLMs
Ansprechpartnerin:
Dr. Layla Tabea Riemann
gerne formlos per E-Mail an: l.riemann@uke.de
Knowledge Integration in Precsion Medicine -
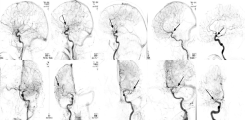
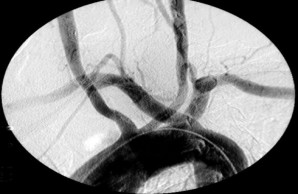
AngioLLM
Studienarbeit: AngioLLM (Bachelor/Projekt/Master)
LLMs und Videos:
Erlernen von Angiographie-Videorepräsentationen aus BefundtextenVideos aus angiographischen Herzkatheteruntersuchungen zeichnen sich durch eine hohe Komplexität aus. Sowohl der zeitliche Verlauf des Kontrastmitteleinflusses in die Gefäße als auch die Kombination von Informationen aus mehreren Blickwinkeln nötig ist, um eine vollständige Beschreibung des Gefäßzustandes zu erhalten. Manuelle Annotationen erfordern Expertenwissen und sind oft auf einzelne Fragestellungen beschränkt. Das Ziel dieses Projekts ist es zu untersuchen, ob die Kombination aus Befundtexten und Videos ohne zusätzliche Expertenannotation geeignet ist, um eine sinnvolle Repräsentation von angiographischen Aufnahmen zu erlernen. Außerdem soll geprüft werden, ob diese Kombination genutzt werden kann, um automatische Annotationen zur Validierung zu extrahieren.
Aufgaben:
- Extraktion von Datenannotationen durch Anwendung von Natural Language Processing (NLP) und Large Language Models (LLMs) auf Befundtexte
- Aufsetzen einer Trainingspipeline zum simultanen Erlernen von Befund- und Videorepräsentationen
- Validierung der Ergebnisse anhand von ICD-10-Codes und anderen relevanten medizinischen Daten
Voraussetzungen:
- Technischer Studiengang wie Informatik/ Physik/ Bioinformatik/ (Medizin-) Ingenieurwesen oder ähnliches
- Grundkenntnisse im (Objekt-orientierten) Programmieren mit Python
- Gerne erste Erfahrungen mit Deep (pytorch) & Machine Learning (scikit-learn)
Ansprechpartner:
Prof. Dr. René Werner,
gerne formlos an r.werner@uke.de
Image Processing and Medical Imaging Group -
Simulation Framelabeling
Studienarbeit: Simulation Framelabeling (Bachelor/Projekt/Master)
Effiziente Annotation medizinischer Videos:
Welche Frames sind wirklich wichtig?Effiziente Annotationsprozesse sind ein wichtiger Pfeiler für den Einsatz von Deep-Learning-Systemen im medizinischen Kontext. Dies gilt insbesondere für die Annotation von Videos, wenn Annotationen für einzelne Frames vorgenommen werden. Obwohl das Encoding der Videoframes grundsätzlich durch Self-Supervision labelfrei erlernt werden kann, sind für eine aufgabenspezifische Prädiktion solche Annotationen unerlässlich.
Ziel der Arbeit ist es, auf Basis einer Deep-Learning-Videoklassifikationspipeline verschiedene Strategien zur Präselektion zu annotierender Einzelframes zu entwickeln und hinsichtlich ihrer Effizienz zu evaluieren. Hierfür steht ein gelabelter Videodatensatz aus Laryngoskopien zur Verfügung. Bei allen Experimenten handelt es sich zunächst um Simulationen.Aufgaben:
- Entwicklung einer Simulationspipeline für sparse annotations (Annotation weniger, aber aufgabenspezifisch sinnvoller Frames)
- Weiterentwicklung einer Trainingspipeline für sparse annotations
- Evaluierung verschiedener Strategien zur optimalen Selektion der Frames (z.B. über die Varianz verschiedener Frames, Optical Flow, Latent Space-Repräsentation, etc.)
Voraussetzungen:
- Technischer Studiengang wie Informatik/ Physik/ Bioinformatik/ (Medizin-) Ingenieurwesen oder ähnliches
- Grundkenntnisse im (Objekt-orientierten) Programmieren mit Python
- Gerne erste Erfahrungen mit Deep (pytorch) & Machine Learning (scikit-learn)
Ansprechpartner:
Prof. Dr. René Werner
gerne formlos an r.werner@uke.de
Image Processing and Medical Imaging -
LLM - Generierung synthetische Daten
Studienarbeit: LLM - Generierung synthetische Daten (Bachelor/Projekt/Master)
LLM-Einsatz zur Generierung von synthetischen, medizinischen Sekundärdaten
Seit Oktober 2022 ist es am UKE möglich, mittels des Datenhotels medizinische Sekundärdaten aus verschiedensten Datenquellen generieren und ausgeben zu lassen, um diese für die Auswertung und Hypothesengenerierung nutzen zu können. Allerdings beschränkt sich die Ausgabe aus Datenschutzgründen auf ein vor Ort gebundenes System. Die Generierung von synthetischen Daten könnte zunächst einen praktischen Zwischenschritt darstellen, um eine externe Analyse gewünschter Kohorten zu ermöglichen.
In diesem Projekt sollen mittels LLMs oder anderen Architekturen synthetische Daten für die externe Nutzung generiert werden. Dabei soll die statistische und pragmatische Nutzbarkeit dieser Modelle auf verschiedenen Datensätzen des Datenhotels evaluiert werden.Aufgaben:
- Einsatz von LLMs zur Generierung von realistischen synthetischen Daten auf Grundlage der Daten des Datenhotels
- Entwicklung einer semi-automatischen Pipeline für variable Datensätze
- Validierung der Ergebnisse anhand statistischer und „Utility“ Metriken (Fragestellungen auf den Datensätzen)
Voraussetzungen:
- Technischer Studiengang wie Physik / Bioinformatik / Informatik / (Medizin-) Ingenieurwesen oder ähnliches
- Grundkenntnisse Jupyterlab und Python
- Wünschenswert: Erste Erfahrungen mit dem Einsatz von LLMs
Ansprechpartnerin:
Dr. Layla Tabea Riemann
gerne formlos per E-Mail an: l.riemann@uke.de
Knowledge Integration in Precision Medicine -
Videolabeling Pipeline
Medizinische Promotion oder Studienarbeit: Videolabeling Pipline (Bachelor/Projekt)
Entwicklung eines Human-in-the-Loop-Systems zur KI-basierten Klassifikation von Laryngoskopievideos
Deep-Learning-Lösungen für das Erlernen eines Verständnisses und die Klassifikation von Sequenzen oder einzelnen Frames medizinischer Videos wie z.B. Laryngoskopievideos stellen aus zweierlei Gründen eine Herausforderung in der automatischen Bildverarbeitung dar:
1.) das erforderliche Annotieren von Einzelframes ist zeitaufwändig und erfordert Expertenwissen
2.) das Training von Deep-Learning-Modelle benötigt eine große Anzahl annotierter Datenpunkte
Durch den Einsatz von nicht annotierten Daten lässt sich durch selbstüberwachtes Lernen die Anzahl der zur Klassifikation benötigten Daten zwar reduzieren, der Bedarf einer effizienten Pipeline zur Annotation bleibt jedoch weiterhin bestehen. Ziel dieser Arbeit ist es, eine effiziente Annotations-Pipeline auf der Basis von CVAT (Computer Vision Annotation Tool) aufzusetzen und verschiedene Szenarien wie das Labeln von einzelnen Frames oder ganzen Videoabschnitten zu untersuchen. Außerdem soll eine effizienter Human-in-the-Loop-Ansatz implementiert werden, der ein kontinuierliches Online-Training von Klassifikationsmodellen ermöglicht.Aufgaben:
- Entwicklung einer Annotationspipeline mit CVAT mit folgenden Anforderungen:
- Annotation relevanter Einzelframes und zusammenhängender Videoabschnitte
- Möglichkeit der Erstellung eines Konsensus-Ratings
- Weiterentwicklung der von Deep Learning-Trainingspipelines für einen Human-in-the-Loop-Betrieb
- Evaluation von Interrater-Reliabilität und Konsensusrating für ausgewählte medizinische Fragestellungen auf Basis von Laryngoskopievideos
- Evaluation des kontinuierlichem Trainings
Voraussetzungen:
- Medizinstudium mit starker technischer Affinität oder Technischer Studiengang wie Informatik/ Physik/ Bioinformatik/ (Medizin-) Ingenieurwesen
- Grundkenntnisse im (Objekt-orientierten) Programmieren mit Python
- Gerne erste Erfahrungen mit Deep (pytorch) & Machine Learning (scikit-learn)
Ansprechpartner:
Prof. Dr. René Werner
gerne formlos an r.werner@uke.de
Image Processing and Medical Imaging Group - Entwicklung einer Annotationspipeline mit CVAT mit folgenden Anforderungen:




_teaserbild.png)